La bioinformática como lanzadera del ARN circular
La Biología Molecular es ese videojuego de ordenador interminable donde tras cada nivel aparecen personajes nuevos y misiones aparentemente complejas. El último parche (término que se utiliza coloquialmente cuando se actualiza un videojuego) incluye un nuevo objetivo: el ARN circular (circRNA). Hasta el momento, la bioinformática es la principal vía para tratar de entender estas moléculas.
El ARN circular es una cadena de ácido ribonucleico en forma de circuito cerrado. Este tipo de moléculas fueron descubiertas por primera vez hace 40 años en viroides vegetales, pero no fue hasta la década de los 90 cuando se encontró el primer ARN circular en humanos. Desde entonces se empezó a cocer a fuego lento una serie de herramientas y técnicas de laboratorio con el fin de determinar la función biológica de estas moléculas. En los últimos 5 años el interés generado en base al ARN circular ha crecido exponencialmente. Hemos visto cómo esta molécula ha pasado de ser considerada un subproducto del splicing alternativo de ARN a encontrarse vinculada con la regulación en tejidos, en la biogénesis, la degradación, la traducción, el transporte, las interacciones y la conservación evolutiva. Demasiadas tareas para algo que a priori parecía ser un desecho. Pero la enumeración de tareas no basta, varios estudios relacionados con el cáncer le otorgan el galardón de biomarcador, es decir, una característica más que podría ayudar a los clínicos a realizar el diagnóstico, pronóstico y tratamiento de los pacientes con cáncer.
El ARN circular es una cadena de ácido ribonucleico en forma de circuito cerrado. Este tipo de moléculas fueron descubiertas por primera vez hace 40 años en viroides vegetales, pero no fue hasta la década de los 90 cuando se encontró el primer ARN circular en humanos. Desde entonces se empezó a cocer a fuego lento una serie de herramientas y técnicas de laboratorio con el fin de determinar la función biológica de estas moléculas. En los últimos 5 años el interés generado en base al ARN circular ha crecido exponencialmente. Hemos visto cómo esta molécula ha pasado de ser considerada un subproducto del splicing alternativo de ARN a encontrarse vinculada con la regulación en tejidos, en la biogénesis, la degradación, la traducción, el transporte, las interacciones y la conservación evolutiva. Demasiadas tareas para algo que a priori parecía ser un desecho. Pero la enumeración de tareas no basta, varios estudios relacionados con el cáncer le otorgan el galardón de biomarcador, es decir, una característica más que podría ayudar a los clínicos a realizar el diagnóstico, pronóstico y tratamiento de los pacientes con cáncer.
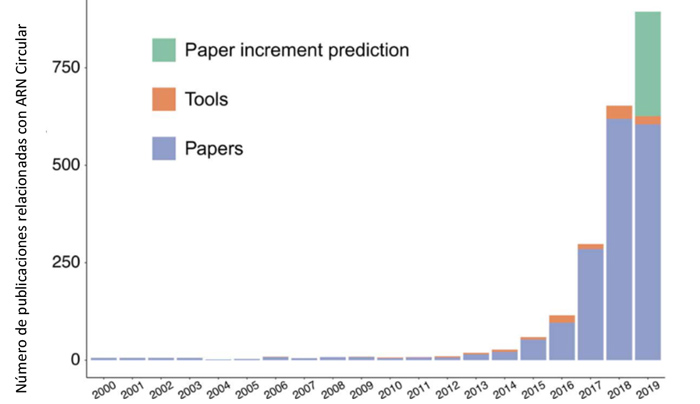
Chen, L. et al. (Briefings in Bioinformatics, 2020)
El quid de la cuestión radica en cómo se pueden identificar los ARN circulares. La bioinformática se ofrece como pieza clave de este rompecabezas. En 2012 se publica el primer software libre para identificar ARN circular y desde entonces se han publicado más de 100 herramientas relacionadas con su identificación y anotación. Para la identificación de ARN circular se emplean los resultados obtenidos de un RNA-seq. Actualmente, nuestro jugador principal con la skin (término coloquial que hace referencia a las armas y apariencia de un personaje en un videojuego) es de bioinformático y podemos diferenciar 3 ataques distintos.
El primero de ellos se basa en la identificación del back spliced junction (BSJ), que se trata del sitio de unión de dos exones. A su vez hay dos estrategias: una basada en pseudorreferencia y otra basada en fragmentos. Mientras que la primera aproximación implica la creación de una pseudorreferencia de uniones exón-exón, el segundo identifica los BSJ a partir de la información de mapeo de la alineación de una lectura con el genoma.
El primero de ellos se basa en la identificación del back spliced junction (BSJ), que se trata del sitio de unión de dos exones. A su vez hay dos estrategias: una basada en pseudorreferencia y otra basada en fragmentos. Mientras que la primera aproximación implica la creación de una pseudorreferencia de uniones exón-exón, el segundo identifica los BSJ a partir de la información de mapeo de la alineación de una lectura con el genoma.
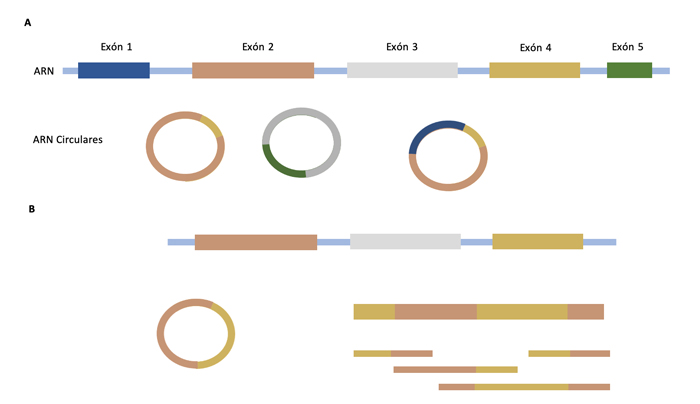
A. Formación de ARN circular a través del Back Spliced Junction (BSJ). B. Identificación de ARN circulares a través de la detección del BSJ.
La segundad habilidad especial recurre a métodos de Machine Learning. La predicción del ARN circular se basa en el empleo del conocimiento de estas moléculas como entrada de un modelo de clasificación. Algunas de esas características son las repeticiones ALU y determinados motivos de secuencias. Entre los diversos algoritmos, podemos encontrar Deep Learning.
Finalmente la última baza juega con la idea explicada en Deep learning y ensemble learning ¿los combinamos?. Se han desarrollado softwares que combinan los resultados de otras herramientas para generar un resultado más robusto y reducir el número de falsos positivos.
Sin embargo, el penúltimo nivel de este videojuego se encuentra incompleto: la estandarización. Se han creado diversas bases de datos que tienen como finalidad recoger los ARN circulares existentes, su funcionalidad, su relación con enfermedades y su vinculación con los microARN. Sin embargo, la falta de una nomenclatura común a estas moléculas dificulta la unificación de la información. Las bases de datos de referencia son circBase y circAtlas para la nomenclatura y CircInteractome para la creación de redes con microARN.
Continuando con la analogía de los videojuegos, la bioinformática es claramente la que se encuentra carrileando (término coloquial para referirse al desempeño extraordinario de un jugador para llevar a la victoria a su equipo) esta partida. La implicación biológica estas moléculas se perfila como una pieza más de este interminable laberinto de conocimiento, no solo en la Biología Molecular, sino también en su potencial impacto en la clínica. Ahora toca pensar: ¿cuál crees que será el próximo nivel?
Finalmente la última baza juega con la idea explicada en Deep learning y ensemble learning ¿los combinamos?. Se han desarrollado softwares que combinan los resultados de otras herramientas para generar un resultado más robusto y reducir el número de falsos positivos.
Sin embargo, el penúltimo nivel de este videojuego se encuentra incompleto: la estandarización. Se han creado diversas bases de datos que tienen como finalidad recoger los ARN circulares existentes, su funcionalidad, su relación con enfermedades y su vinculación con los microARN. Sin embargo, la falta de una nomenclatura común a estas moléculas dificulta la unificación de la información. Las bases de datos de referencia son circBase y circAtlas para la nomenclatura y CircInteractome para la creación de redes con microARN.
Continuando con la analogía de los videojuegos, la bioinformática es claramente la que se encuentra carrileando (término coloquial para referirse al desempeño extraordinario de un jugador para llevar a la victoria a su equipo) esta partida. La implicación biológica estas moléculas se perfila como una pieza más de este interminable laberinto de conocimiento, no solo en la Biología Molecular, sino también en su potencial impacto en la clínica. Ahora toca pensar: ¿cuál crees que será el próximo nivel?
Referencias
1-. Chen, L., Wang, C., Sun, H., Wang, J., Liang, Y., Wang, Y., & Wong, G. (2020). The bioinformatics toolbox for circRNA discovery and analysis. Briefings in Bioinformatics, 00(September 2019), 1–23. https://doi.org/10.1093/bib/bbaa001
Documentación y entrevista
Juan Luis Onieva Zafra para My Scientific Journal 08/11/2020

Juan Luis Onieva ZafraRedactor My Scientific Journal
Bioinformatician & Data Scientist.
